KrakenSmith — pipeline para bancos customizados (Kraken2 & Bracken)
Instalação, construção de bancos, classificação e visualização (Krona).
Sou bioinformata focado em metagenômica, variantes genéticas e automações. Aqui você encontra projetos, tutoriais e um pouco sobre mim.

Bacharel em Ciências Biológicas e Mestre em Ciências (ênfase em genética, mutações e terapia gênica, 2020). Atualmente sou doutorando em Ciências Farmacêuticas pela UNICENTRO, vinculado ao projeto Genomas Paraná. Tenho experiência sólida em genética molecular, citogenética, análises clínicas, terapia celular e medicina regenerativa com células-tronco mesenquimais.
Atuo também na área de bioinformática aplicada à genômica e metagenômica, com foco em análises de microbiomas, anotação de genomas bacterianos e identificação de genes de resistência antimicrobiana. Desenvolvo e aplico pipelines automatizados que abrangem desde o pré-processamento de dados de sequenciamento até montagem, anotação funcional e visualização de genomas, utilizando R, Python e Bash em ambiente Linux.
Tenho ainda familiaridade com bancos de dados relacionais (PostgreSQL, MySQL) e desenvolvimento de soluções web. Sou proficiente no uso do Google Planilhas, explorando recursos avançados para automação, cruzamento e análise de dados de forma visual e integrada.
Instalação, construção de bancos, classificação e visualização (Krona).
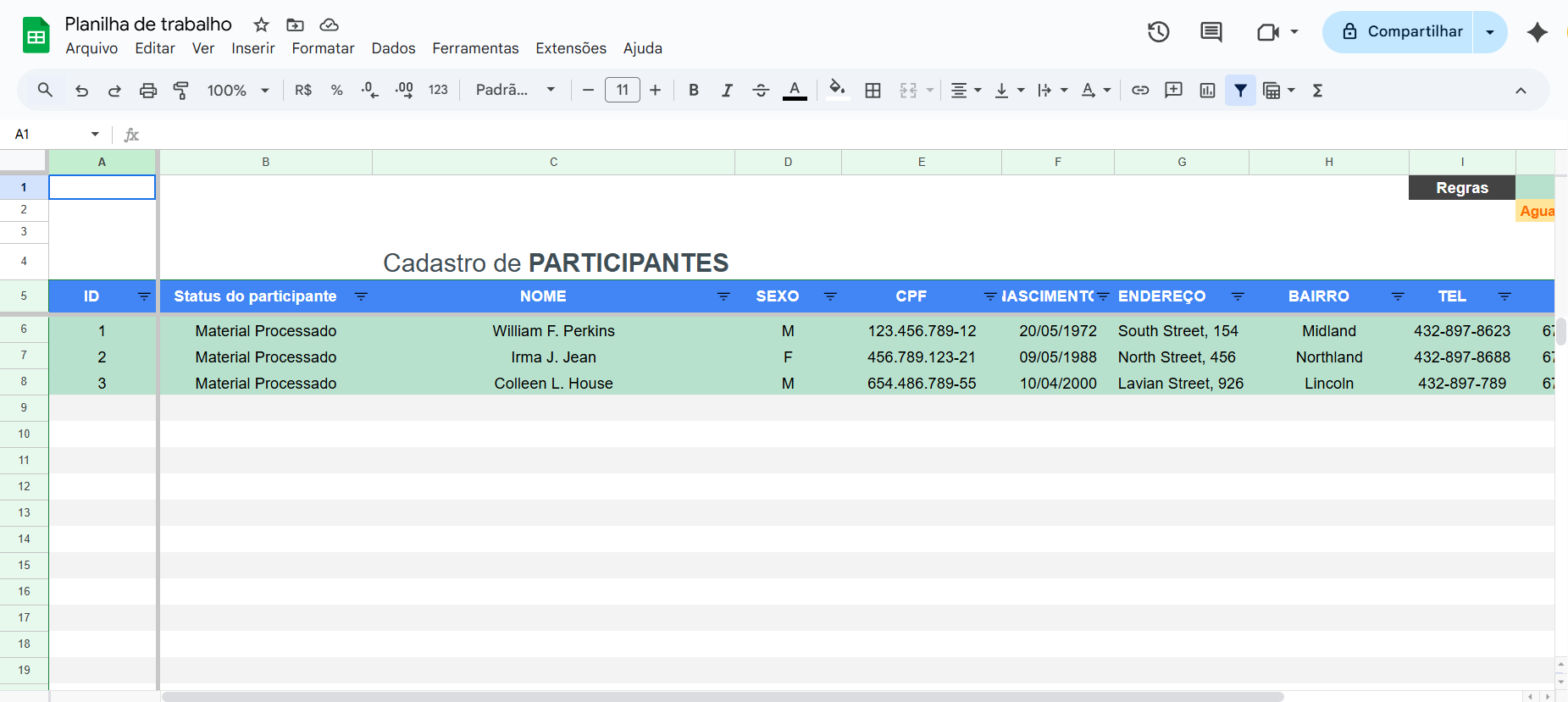
Gerenciamento de cadastros e amostras
Passe o mouse para pausar a rotação automática.
Prefere falar comigo? Envie um e-mail ou conecte-se nas redes.