Tutorial de instalação do SRA-Tools
Tutorial de instalação do SRA-Tools
Fala pessoal, hoje eu vou mostrar pra vocês como instalar o SRA-Tools na máquina.
Quem já tentou baixar dados de sequenciamento do NCBI sabe que não é tão simples assim: clicar em “download” direto do site quase nunca funciona bem, principalmente quando os arquivos são gigantes.
É justamente aí que o SRA-Tools brilha. Ele permite puxar esses dados direto do servidor e já converter no formato certinho pra você usar nas análises.
Eu mesmo já tentei baixar arquivos manualmente e quase sempre dava problema ou quebrava no meio do download, ou vinha num formato meio inútil.
Com o SRA-Tools, você roda um comando simples e pronto, os dados já caem na sua máquina do jeito certo.
Nesse tutorial eu vou te ensinar a instalar o SRA-Tools e fazer o seu primeiro download.
É tranquilo de configurar e, olha... economiza um tempo absurdo quando você precisa trabalhar com dados reais de DNA e RNA.

O que você vai precisar antes:
- Conda — instalação aqui
- WSL/Linux — tutorial aqui
- SRA-Tools — útil para baixar FASTQs públicos (guia)
O que é o SRA-Tools?
O SRA-Tools é um conjunto de ferramentas desenvolvido pelo NCBI que permite baixar, manipular e converter dados do Sequence Read Archive (SRA) Maior repositório público de dados de sequenciamento genômico do mundo. .
Com ele, você consegue pegar dados brutos de DNA/RNA diretamente da base do NCBI e transformar em formatos mais práticos, como FASTQ, para depois analisar em pipelines de bioinformática.
Na prática, o SRA-Tools é tipo a “ponte” entre os dados públicos de sequenciamento e a sua análise local. Seja para montar genomas, rodar metagenômica, RNA-Seq ou testar pipelines, ele é sempre o primeiro passo quando o assunto é buscar dados reais.
Passo a passo
1. Baixando
Antes de começar, você vai precisar do kit básico de sobrevivência do bioinformata: ter o Bioconda instalado. Se estiver no Windows, será necessário trabalhar com o WSL. Os tutoriais estão disponíveis nesses links: WSL e Miniconda.
Tendo o WSL e o Miniconda instalados, a gente pode prosseguir para a instalação.
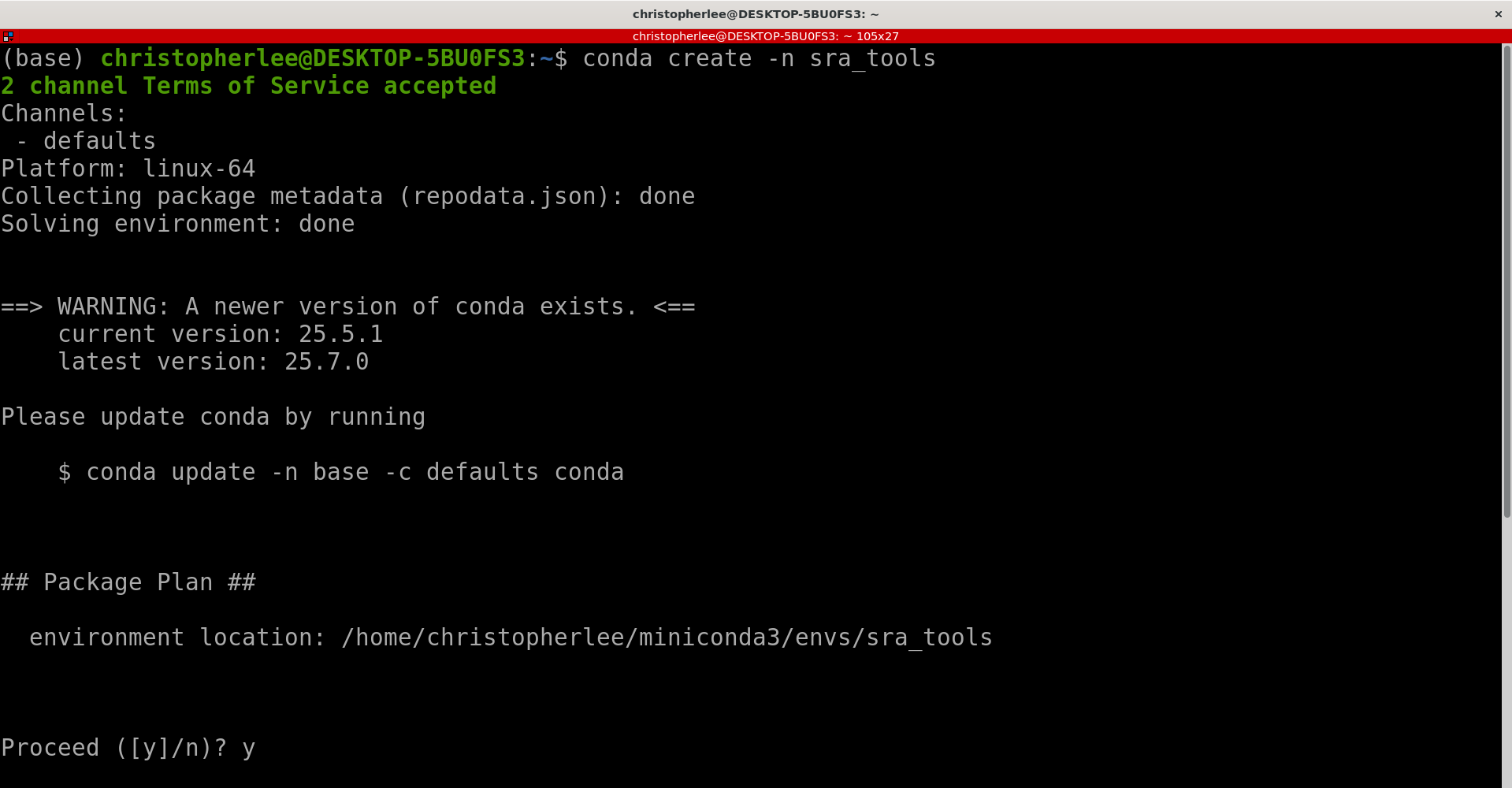
Crie um ambiente no Conda:
conda create -n (nome_do_seu_ambiente)
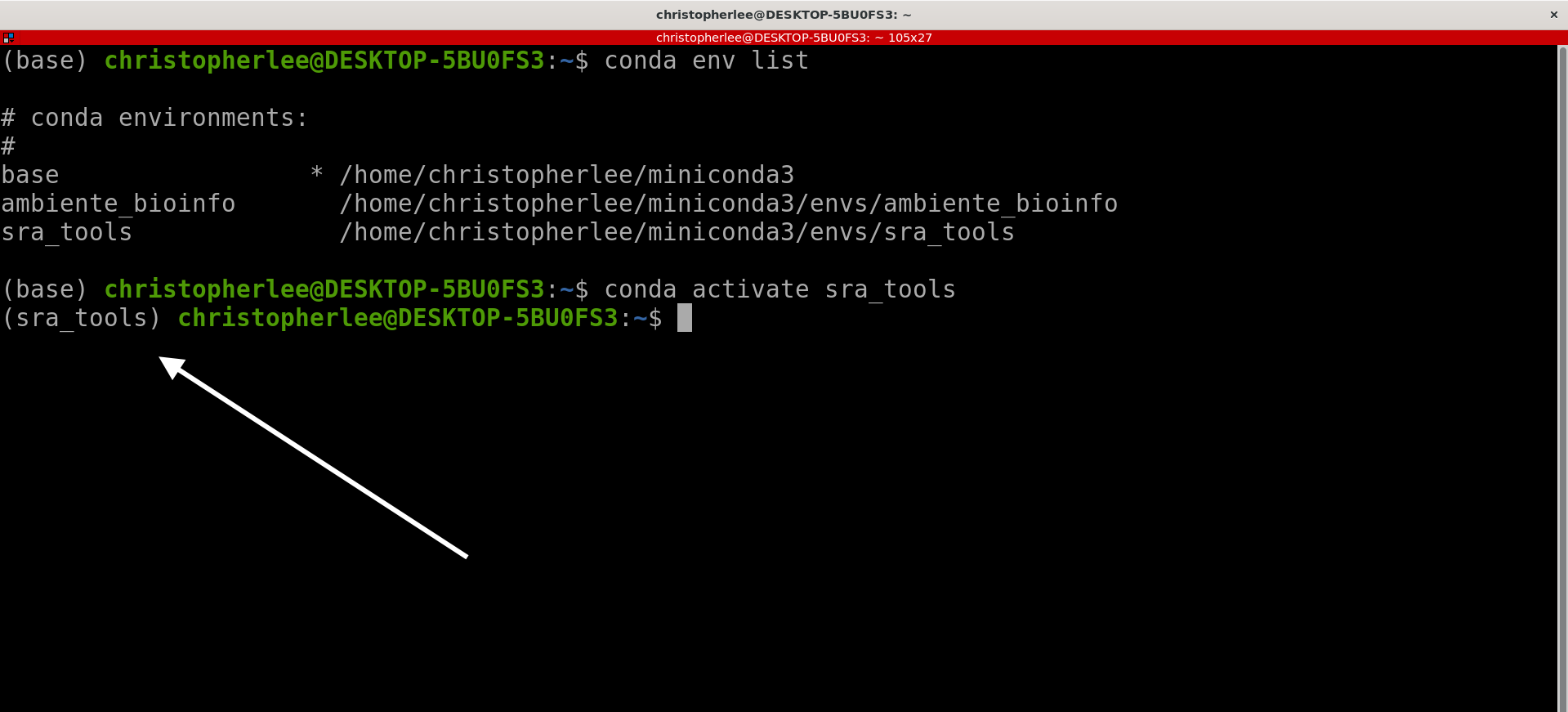
Ative o ambiente criado:
conda activate (nome_do_seu_ambiente)
O que é o Bioconda?
O Bioconda é um repositório de pacotes do Conda voltado para bioinformática. Ele reúne milhares de ferramentas já compiladas e prontas para instalar com um simples comando, sem complicação de dependências. É a forma mais prática de montar ambientes de análise em biociência.
Para baixar um pacote do Bioconda, basta acessar o repositório desejado e usar o comando indicado.
2. Instalando
Instale o SRA-Tools no ambiente ativo:
conda install bioconda::sra-tools
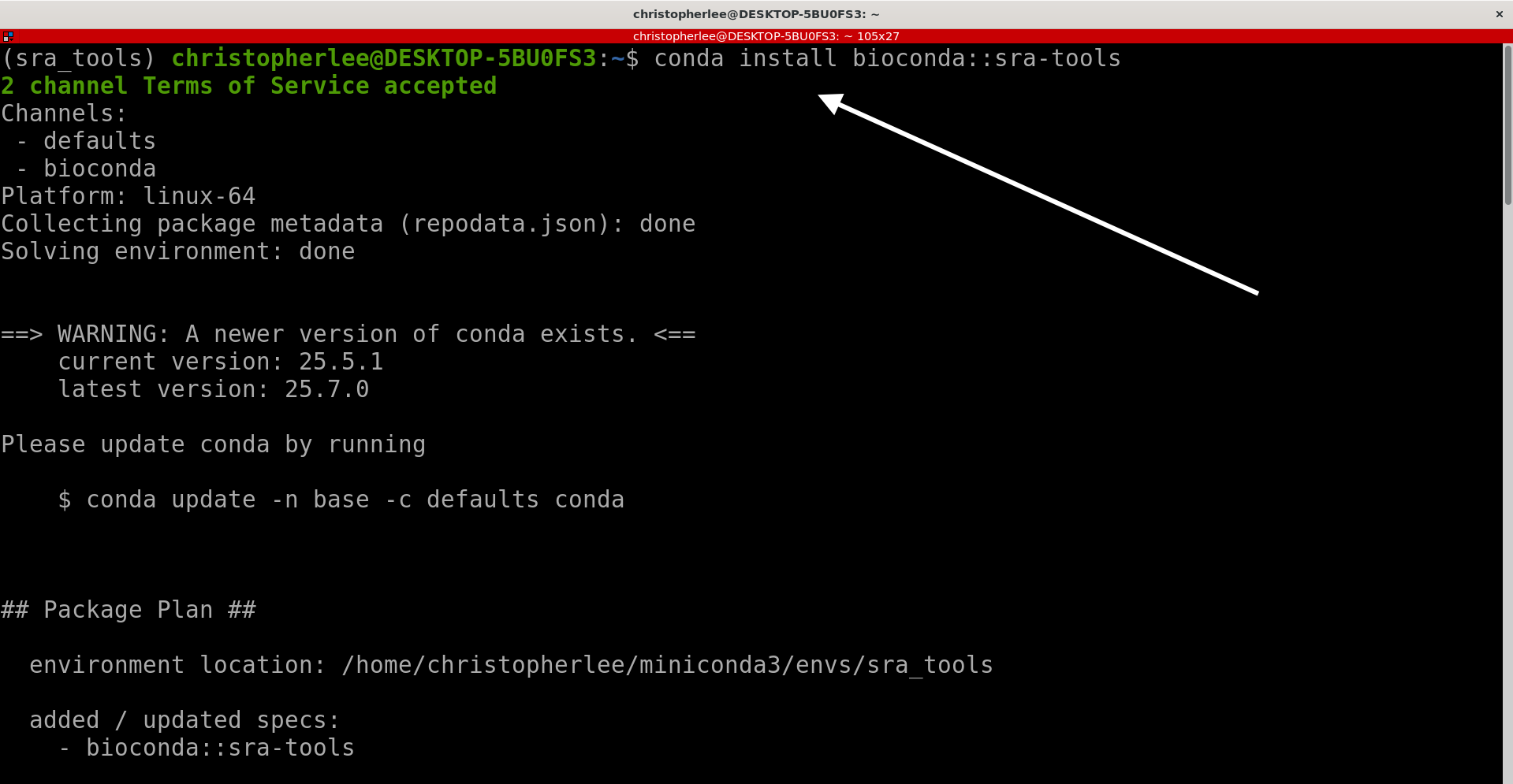
Pronto! O seu SRA-Tools está instalado!
3. Procurando arquivos no SRA
Agora com o ambiente configurado, visite o site oficial do
SRA (NCBI).
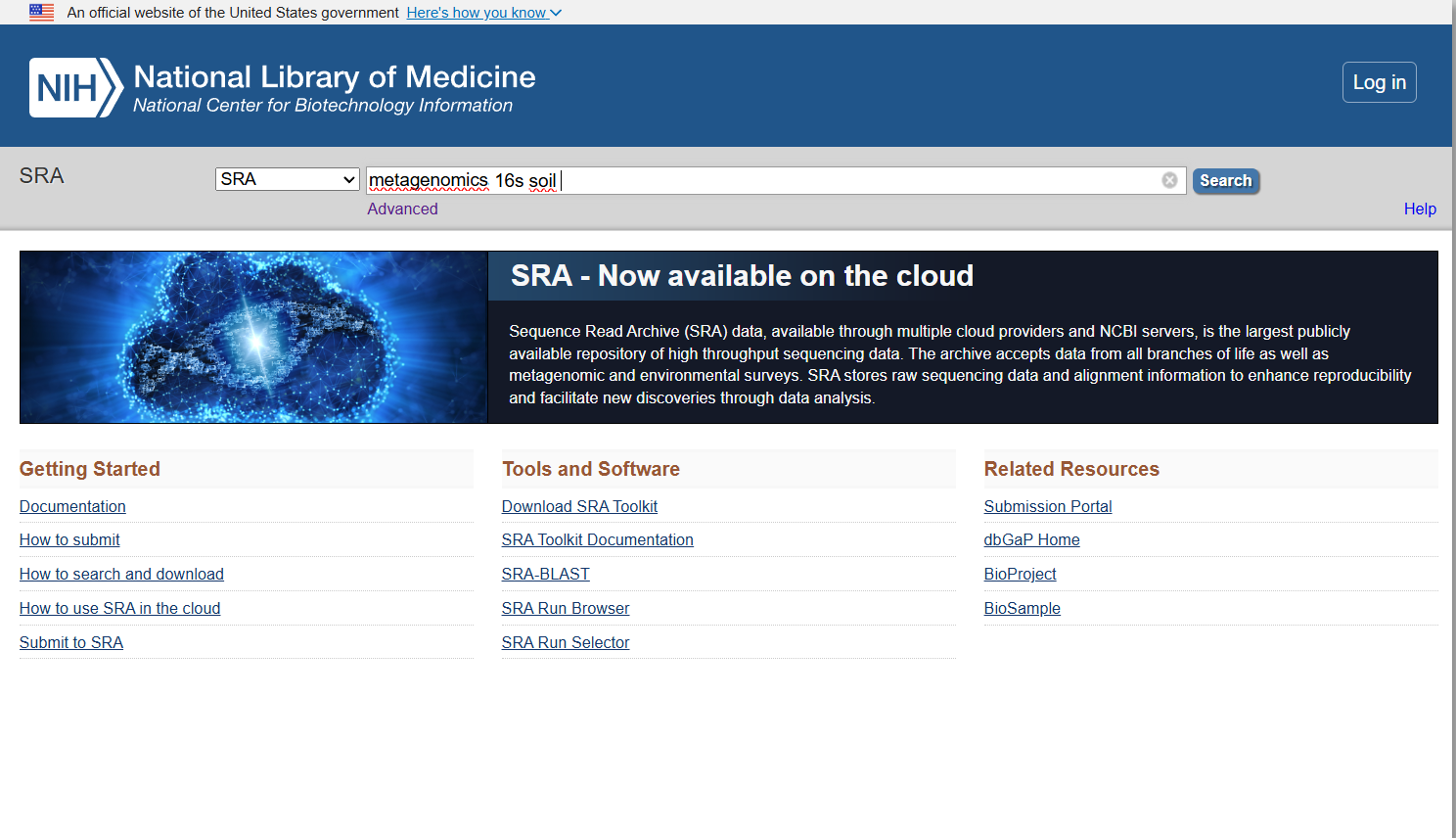
No campo de pesquisa, procure por algo como metagenomics 16S soil.
Você pode usar sua criatividade para procurar outros tipos de estudo: metagenomic 16S gut, metagenomic shotgun gut, transcriptomic, etc.
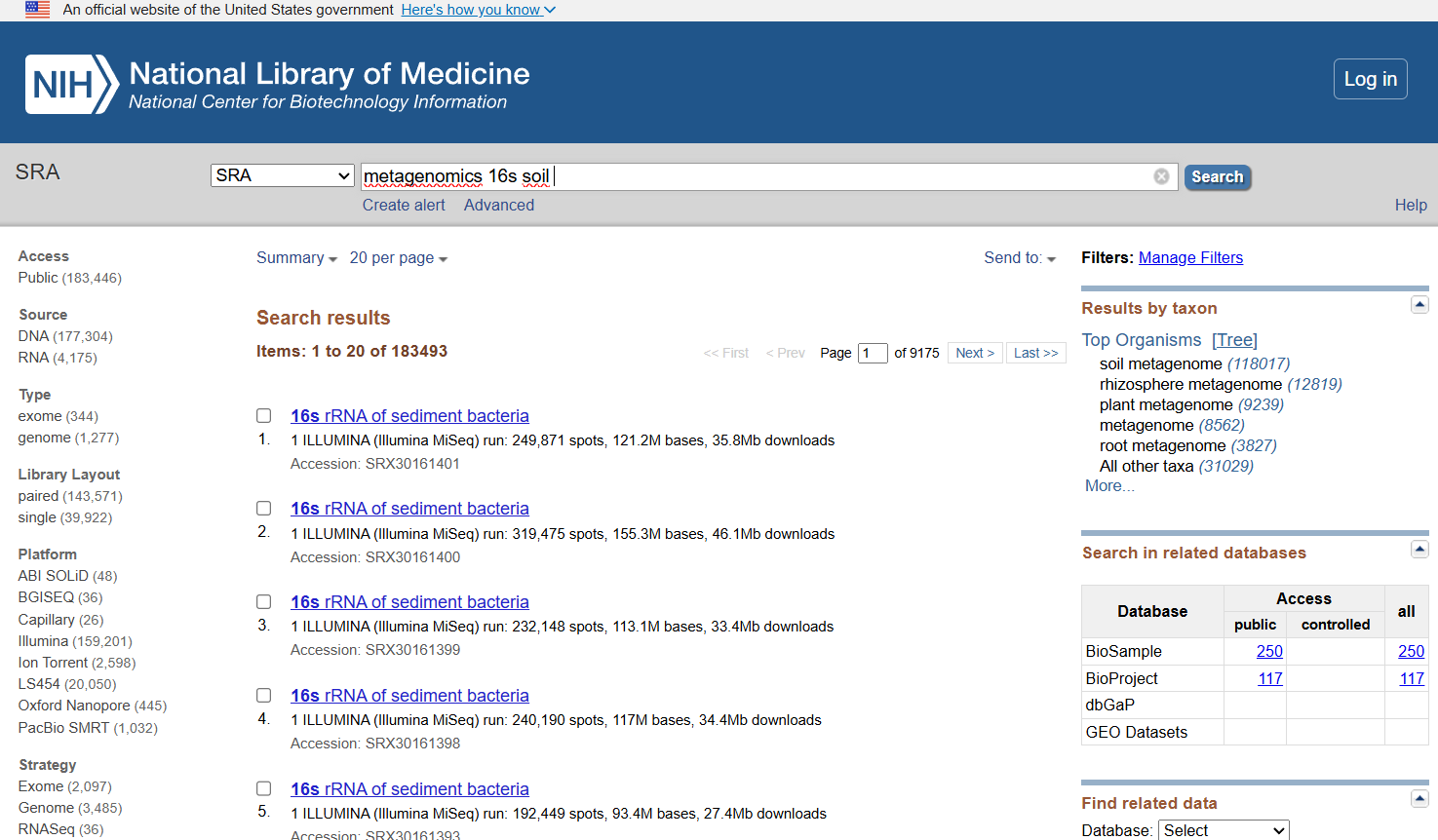
Para fins práticos, escolha um resultado pequeno (ex.: 36 MB).
Não se preocupe se o mesmo estudo não aparecer para você, pode usar qualquer um leve.
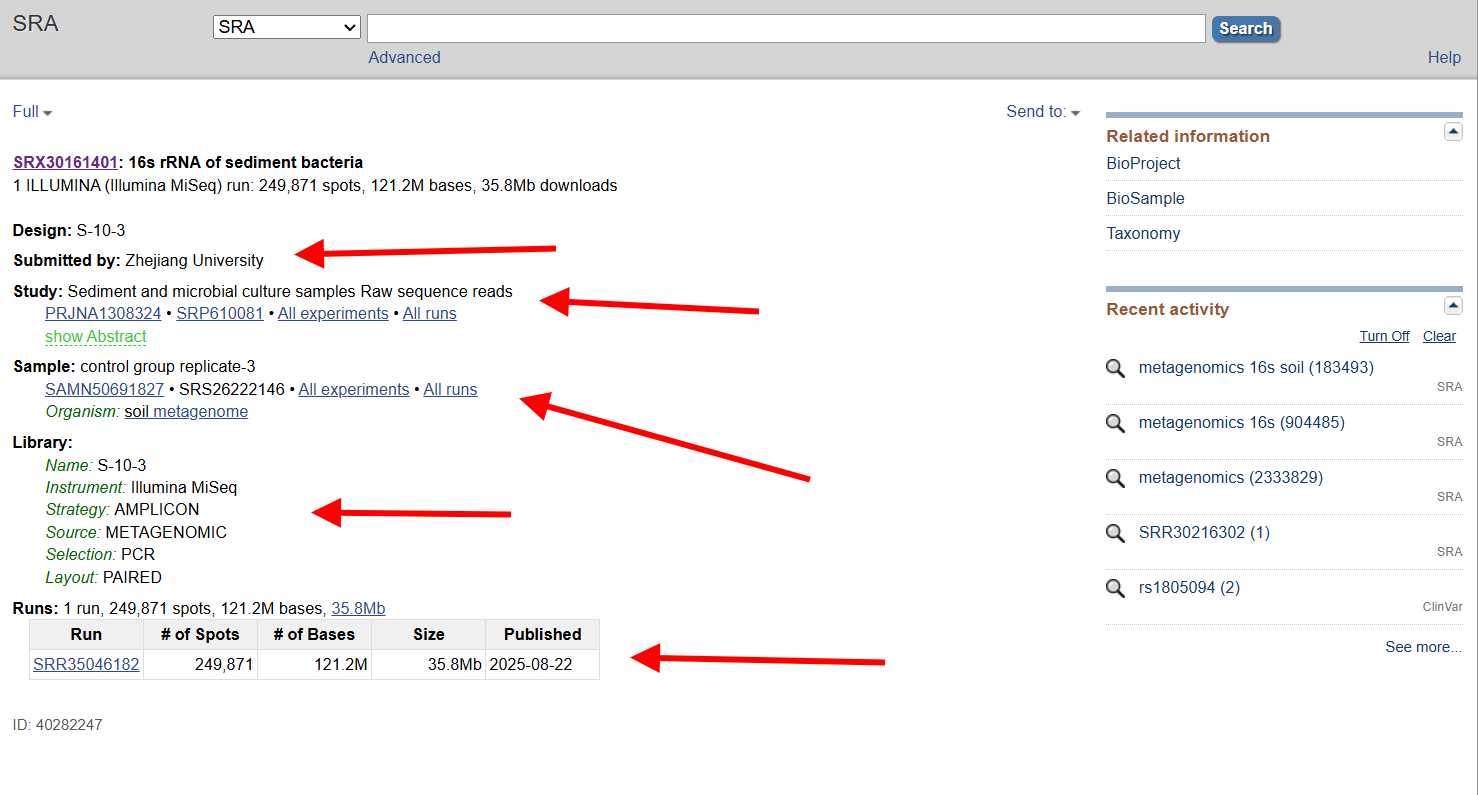
Ao clicar em RUN, você verá os detalhes da amostra.
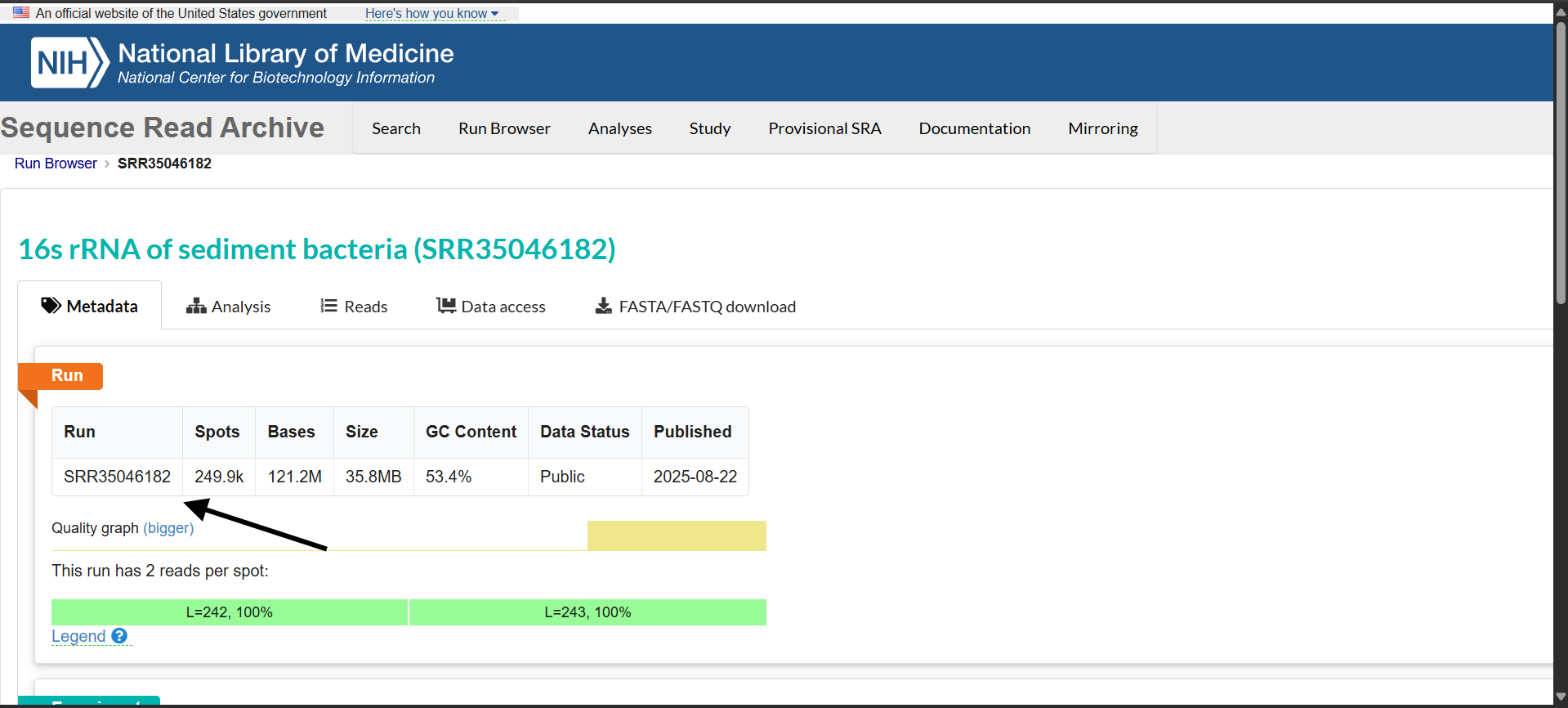
Copie o código da corrida (ex.: SRR35046182).
4. O kit básico que você precisa para sobreviver ao SRA-Tools
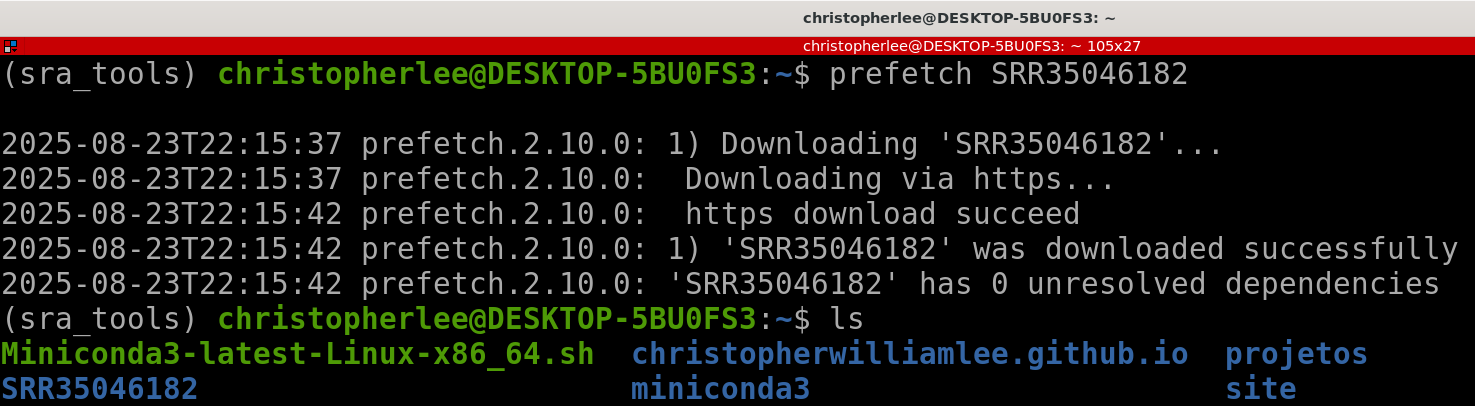
Baixe o arquivo usando o comando:
prefetch SRR35046182
Versões antigas doSRA-Tools(ex.: 2.9.x) podem gerar erros de conexão TLS/SSL ao usarprefetchoufastq-dump. Antes de usar, confira sua versão com:Para desinstalar a versão antiga basta digitar:prefetch -VPara instalar a versão mais nova no mesmo ambiente basta digitar:conda remove sra-toolsconda install -c bioconda -c conda-forge sra-tools -y
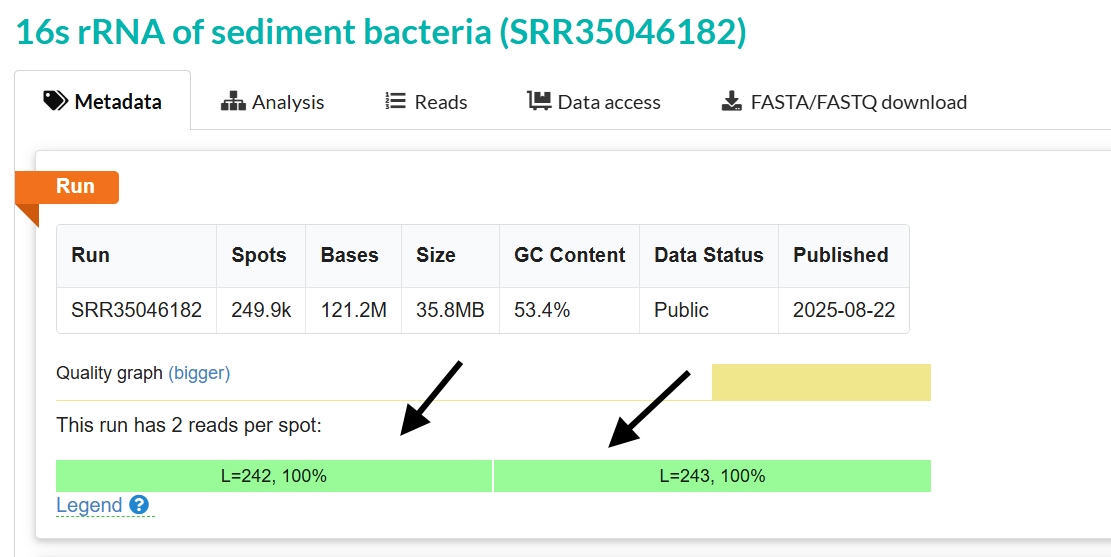
Se a amostra for paired-end, use o comando para separar os arquivos:
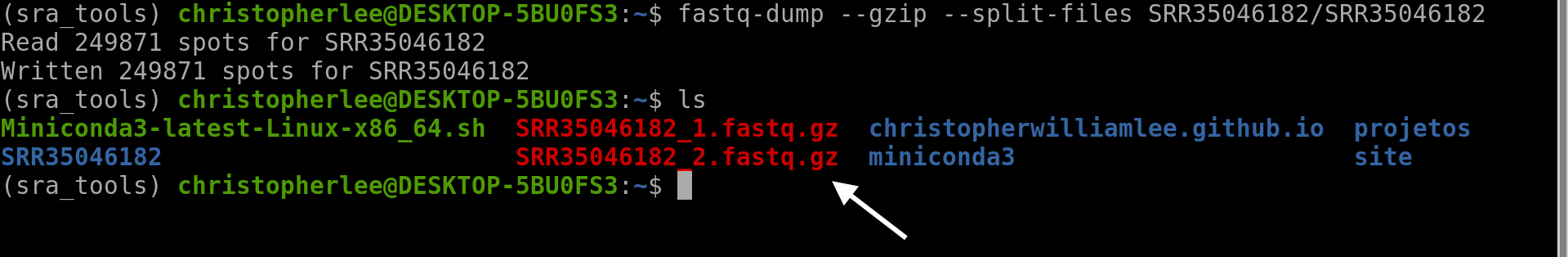
fastq-dump --gzip --split-files SRR35046182/SRR35046182
Pronto! Agora que você tem os arquivos descompactados e separados, pode começar a fazer a análise com as ferramentas de controle de qualidade FastQC, Fastp e Falco.

Fala sério! Esse tutorial é bem mais tranquilo que os outros, né? Viu como a bioinformática não é um bicho de sete cabeças? Brincadeira... às vezes é sim 😅, mas nada que a gente não possa resolver! O segredo é não desanimar e estudar sempre. Stay focus!!
Dê um suporte ao meu projeto. Doe um cafézinho ☕.
Pix: biologolee@gmail.com
Bitcoin: bc1qg7qrfhclzt3sm60en53qv8fmwpuacfaxt5v55k
